NGS解析ソフトウェア
「Amelieff Quick Start Package」
オープンソースソフトウェアやRの実行、解析プロトコルと解析結果の記録まで、1つの環境でデータ解析が完結。 Amelieff Quick Start Packageは、データ解析の効率化に貢献します。
ラインナップ
| ライセンス | 詳細 | 価格 | 納期 |
| 最新 永続版 PC用 ライセンス |
PC1台に解析プロトコル1本を導入致します。 ・ローカルマシンとしてご利用いただくライセンスです。 ※リモート接続非対応 |
¥250,000 (税込 ¥275,000) |
2ヶ月 |
| 最新 永続版 サーバ用 ライセンス |
サーバへセットアップ致します。 ・複数人でリモート接続しご利用いただくライセンスです。 ※弊社製サーバーへの導入時の価格となります。 |
¥600,000~ (税込¥660,000) |
2ヶ月 |
| 解析 システム エントリー モデル |
解析プロトコル1本をノートPCへインストールしてご提供致します。 ノートPCのスペック ・CPU: Core i7 4 core 8 thread ・メモリ: 64 GB ・SSD: 2TB |
¥600,000~ (税込¥660,000) |
2ヶ月 |
解析プロトコル
| 解析プロトコル | 概要 |
| 導入ツール | |
| シングルセル RNAseq解析 |
・発現マトリクスデータから、Seurat を用いた細胞クラスタリング・細胞分類 ・monocleを用いた疑似系譜解析 |
| Seurat, ggplot2, EnhancedVolcano, ReactomePA, clusterprofiler, monocle3, jupyter lab, R, python, Docker | |
| RNAseq解析 | ・QCからリファレンスゲノムへのマッピング ・発現定量、主成分分析、階層的クラスタリング ・発現変動遺伝子の検出、二群間の発現比較結果の可視化 ・パスウェイエンリッチメント解析、GOエンリッチメント解析 |
| FastQC, PRINSEQ, trimmomatic, STAR, featureCounts, gplots, edgeR, EnhancedVolcano, ReactomePA, clusterprofiler, jupyter lab, R, python, Docker | |
| WES 遺伝性変異解析 |
・WESデータを用いたReseq解析 ・QCからリファレンスゲノムへのマッピング、重複リードの除去 ・変異検出、フィルタリング、アノテーション ・IGVによる可視化 |
| FastQC, BWA, GATK, SnpEff, Trimmomatic, IGV, jupyter lab, R, python, Docker | |
| WES がん変異解析 |
・QCからリファレンスゲノムへのマッピング、重複リードの除去 ・正常細胞・腫瘍細胞からの体細胞変異検出、フィルタリング、アノテーション ・oncoplot、lolipop plotによる可視化を中心としたがん体細胞変異の可視化 |
| FastQC,Trimmomatic, BWA, GATK Mutect2, SnpEff, IGV, VEP, maftools(oncoplot, lollipopPlot), SomaticSignatures, jupyter lab, R, python, Docker | |
| ATAC-seq解析 | ・ピーク検出、ピークアノテーション ・Coverage plot、TSS Average profile plot ・パスウェイ解析、結合モチーフ探索 |
| FastQC, Trimmomatic, Bowtie2, Samtools, MACS2, ChIPseeker, HOMER, jupyter lab, R, python, Docker | |
| 細菌叢解析 | ・Qiime2によるメタゲノム解析 ・クオリティコントロール ・系統樹作成、多様性解析(α多様性の検定、β多様性の検定) ・taxon解析 |
| Qiime2,jupyter lab, R, python, Docker |
【システム要件】 CPU: 4core以上、メモリ: 64GB以上、ディスク: 2TB以上
必要システム構成
・Windows 10 (64-bit) / macOS 10.14 Mojave 以降・4 core以上のCPU
・64GB以上の RAM(最低16GB以上)
・1TB以上のHDD
※必要システム構成は解析プロトコルによって異なります。Amelieff Quick Start Packageインストール済みの「Amelieff Quick Start Package解析システム」もご提供しておりますのでお問い合わせください。
※Docker For Windows、Docker for Macなどの利用にかかるライセンス(契約形態により有料)は本製品とは別でご購入いただく必要がございます。
特徴
Amelieff Quick Start Packageは、PCに専用のソフトウェアをインストールして利用するデータ解析支援システムです。
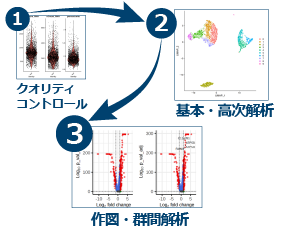
■ 検証済みの解析プロトコルを用いて、データ解析を自動で実行
研究論文を参考に組み上げた検証済みのアメリエフ独自の解析プロトコルをご提供します。 ベーシックな解析に加え、解釈できる結果を得るための高次解析まで、一連のデータ解析を自動で実行することができます。 スタンダードな解析はすぐに実行することができます。 解析の進捗、結果結果の図表は、操作画面にリアルタイムで表示されます。 トレンドの解析を、再現・検証・導入するハードルを下げます。
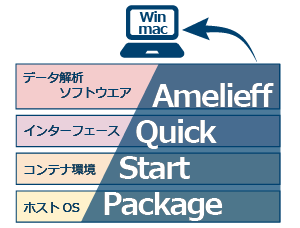
■ Linux,Rを用いた実行から記録まで、解析環境を1つに集約
R、Python、Linux、Jupyter notebook、解析に必要な各種オープンソースソフトウェアをパッケージ化いたしました。 これまでは、Linuxサーバに加え、解析用のパソコンを準備するのが一般的で、使用するソフトウェアに合わせて解析環境を変えたり、ファイルを移動させたり、解析環境毎にプロトコルを用意する必要がありました。 このパッケージを利用すると、解析に必要な環境やソフトウェアを複数用意する必要はありません。ひとつのノートブックから解析を実行する感覚で解析を行えます。
■ データ解析の柔軟な拡張性と再現性の確保
データ解析の過程で必要になった、手順のブラッシュアップや新しい手法の追加などをプラットフォームに縛られず行うことができます。また、実行した手順は記録に残るため、実行した解析環境と手順、解析の意図や結果まですべてのノウハウを保存・再現できます。 解析を進める過程や、論文が投稿され受理されるまでの期間、複数バージョンの解析環境を維持・保存することができます。 使用方法に加え、解析のお困りごとまでしっかりサポートいたします。


