事前学習併用型トレーニング
動画視聴とハンズオン質問会を組み合わせた事前学習併用型トレーニングを提供いたします。
平日の日中に時間が取れない方、隙間時間に学習したいに方に、最適なプランをご提供します。

・バイオインフォマティクスの経験豊富な講師によるトレーニング動画を視聴(2週間)
・動画視聴期間はトレーニング用サーバを利用できるので自分のペースで実践的な学習が可能
・事前学習終了後に復習や疑問解消のための Web会議形式のハンズオン質問会を開催
【ハンズオン質問会のイメージ画像】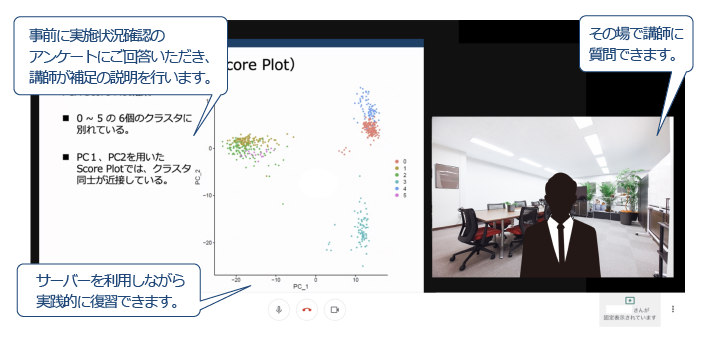
【事前学習併用型トレーニング対応科目】
- 基礎科目CLOSE
-
Linux入門 学習目的 多くの解析の基盤となるLinux操作技術を、バイオインフォマティクスに必要十分なレベルで習得 講義内容 Windows, Macとは異なるOSのディレクトリ構造の理解、テキストファイル加工、ソフトウェアインストールなど R入門 学習目的 バイオインフォマティクス業界で頻繁に使われる統計ソフト Rの操作技術習得 講義内容 Rの基本的な操作方法、作図、解析目的に応じた公開パッケージのインストール・使用方法など
- 応用科目CLOSE
-
RNA-seq解析 学習目的 RNA-seq解析による発現変動遺伝子の検出、二群間比較の習得 講義内容 公開シーケンスデータを用いたオープンソースソフトウエアによる、QCからGO・パスウェイエンリッチメント解析までの実践的な実習 シングルセル
RNA-seq解析
【Seurat】学習目的 Seuratを用いたシングルセルRNA-seq解析技術を習得 講義内容 公開シーケンスデータを用いたオープンソースソフトウエアによる、クラスタリングやマーカー遺伝子探索とセルタイプ同定などの実践的な実習 シングルセル
RNA-seq解析
発展学習目的 シングルセルRNA-seq解析の擬似系譜解析とvelocity解析を習得 講義内容 公開シーケンスデータを用いたオープンソースソフトウエアによる、疑似系譜解析・RNA velocity解析の実践的な実習
豊富な応用科目からご選択いただける「同時双方向型トレーニング」も併せてご検討ください!
お客様の声
事前学習期間中に自分のペースで、理解度の浅いところなど重点的に学習できた。また、整備されたサーバ上でデータ解析を繰り返し実習でき、トライ&エラーで理解度が上がってよかった。事前学習でつまづいた部分もあったが、ハンズオン質問会で解決できた。
大学臨床研究医

平日の昼間は皆忙しく、突発的なオペがあったりと都合も合わせづらいので、バラバラの日程でも受講ができる事前学習併用型を選んだ。おかげで日曜、祝日、夜間など自分の都合の良い時間に学習できた。
大学臨床研究医

ハンズオン質問会ではデータ解析の単純な解説にとどまらず、妥当なパラメータ設定やどういう場合に役立つツールかなど実践を想定した疑問について、専門の方から有益なことを教えてもらえたので大変良かった。
国立研究所研究員

解析ソフトウエア
事前学習併用型トレーニングとQuick Start Packageを組み合わせることで、ご自身の研究のデータ解析をシームレスにはじめていただけます。

- メリット
- ・論文調査からご自身の研究データを用いた解析まで、最短2ヶ月で解析をはじめられる
- ・解析環境構築(コンピュータ準備/ツールのインストール/ワークフローの自動化)に悩まされることがない
- ・パラメータのチューニングなどトレーニング内容を踏まえ、自分で解析をカスタマイズできる
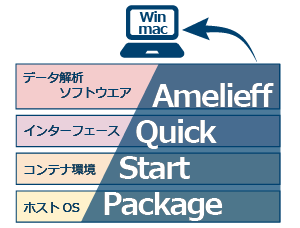
- NGS解析ソフトウェア「Amelieff Quick Start Package」詳細OPEN
-
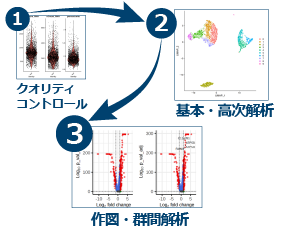
オープンソースソフトウェアやRの実行、解析プロトコルと解析結果の記録まで、1つの環境でデータ解析が完結。Amelieff Quick Start Packageは、データ解析の効率化に貢献します。
【特徴】
検証済みの解析プロトコル
解析環境を1つに集約
拡張性と再現性の確保

検証済みの解析プロトコル

解析環境を1つに集約

拡張性と再現性の確保

【ラインナップ】
解析プロトコル1種類(RNAseq解析、シングルセルRNAseq解析)をセットアップいたします。
ライセンス 詳細 価格 納期 最新 永続版
PC用
ライセンスPC1台に解析プロトコル1本を導入致します。
・ローカルマシンとしてご利用いただくライセンスです。
※リモート接続非対応¥250,000
(税込 ¥275,000)2ヶ月 最新 永続版
サーバ用
ライセンスサーバへセットアップ致します。
・複数人でリモート接続しご利用いただくライセンスです。
※弊社製サーバーへの導入時の価格となります。¥600,000~
(税込¥660,000)2ヶ月 解析
システム
エントリー
モデル解析プロトコル1本をノートPCへインストールしてご提供致します。
ノートPCのスペック
・CPU: Core i7 4 core 8 thread
・メモリ: 64 GB
・SSD: 2TB¥600,000~
(税込¥660,000)2ヶ月


