シングルセルRNA-seq解析ソフトウエア
「Amelieff Quick Start Package」には、R、Linux、シングルセルRNA解析に必要なソフトウェア環境がセットアップされています。 発現マトリクスデータから、Seurat を用いた細胞クラスタリング・細胞分類、monocleを用いた擬似系譜解析まで実行できます。 複数の論文に基づき作成し複数のデータで検証した解析手順をご使用いただけます。
シングルセル解析の
データ統合と
統合後の群間比較を
自動化できる解析ソフトウエア
25万円~
(税込275,000円~)
※PC用永続版ライセンスの場合
特徴
Amelieff Quick Start Packageは、シングルセルRNA-seq解析でご要望の多いデータ統合と統合後の群間比較といった複雑なステップを、タッチポイントの少ないシンプルな操作のみで実行することができます。
【シングルセルRNA-seq解析におけるデータ統合とは】
シングルセルRNA-seq解析の場合、実験条件の影響を受けやすく単純に比較解析することができません。複数のシングルセルデータをバッチエフェクト補正した上で統合することで、異なる研究の結果を比較し、新しい洞察を得ることができます。
統合されたデータは、細胞のクラスタリングや分類を行うことができます。これにより、異なるサンプル間で類似性の高い細胞群を識別し、発現変動の大きい遺伝子や細胞の種類を特定できます。
【簡便なシングルセルRNA-seq解析ソフトウェア】
シングルセルRNA-seq解析で一般的に使用されるSeuratでは、さまざまな関数を呼び出し、データを加工しながら解析を進める必要があるため、データ構造やアルゴリズムに対する理解が求められます。Amelieff Quick Start Packageでは、クラスタリングやデータ統合などのステップを簡単なパラメータ設定のみで自動的に実行できるため、どなたでも簡単にシングルセルRNA-seq解析を行うことが可能です。また、解析経験が豊富なバイオインフォマティストによるパラメータ設定が組み込まれているため、Seuratチュートリアルよりも最適化されたシングルセルRNA-seq解析を実行できます。
機能一覧
| 機能 | 詳細 | |
| 1 | QC | ・集計・可視化 ・低クオリティ遺伝子および細胞の除外 |
| 2 | 次元削減とクラスタリング | ・次元削減(PCA、UMAP) ・クラスタリングおよびUMAP上の可視化 |
| 3 | 各クラスターにおける高発現遺伝子の同定 | ・クラスター毎に発現が異なる遺伝子を探索 ・細胞腫に特異的に発現するマーカーからクラスターを識別 |
| 4 | Feature plot 作図 | ・着目遺伝子の発現量を次元圧縮図上にプロットし可視化 |
| 5 | 機能解析 | ・Gene Ontology解析 ・パスウェイ解析 ・擬似系譜解析 ・データ統合 ・全クラスターにおける2群間発現比較 ・オブジェクト保存 |
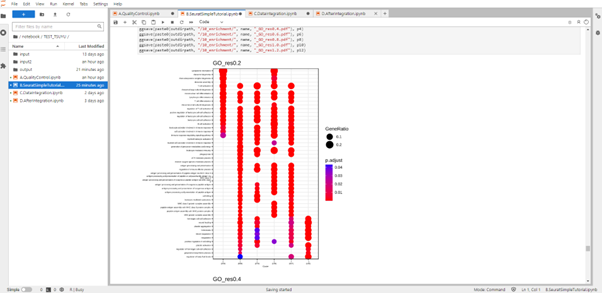
Gene Ontology解析
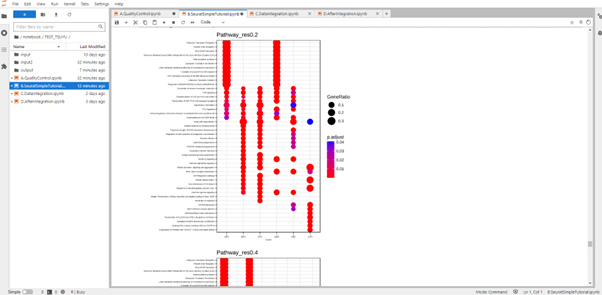
パスウェイ解析
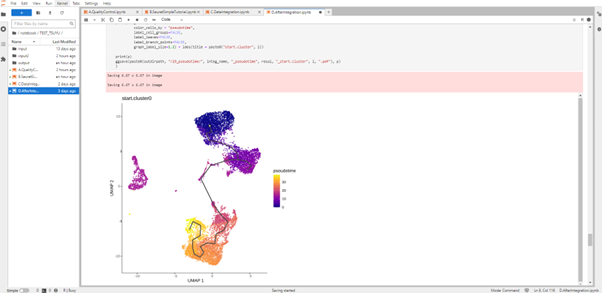
擬似系譜解析
データ統合
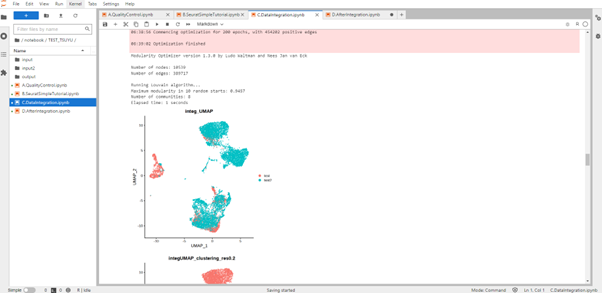
全クラスターにおける2群間発現比較
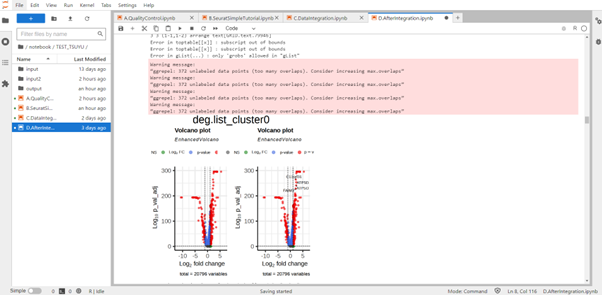





その他
| 必要PCスペック | CPU : 4 core メモリ : 64 GB HDD : 2TB |
| 導入ツール | Seurat, EnhancedVolcano, ReactomePA, monocle3, jupyter lab, R, Docker |
| 入力データ | 10x Cell Ranger 発現マトリクス ※シングルセルレパトア解析データには対応していません。 |
| 対応生物種 | ヒト、マウス、ラット |

