amelieff home > 空間的遺伝子発現解析ソフトウエア
空間的遺伝子発現解析ソフトウエア
「Amelieff Quick Start Package」には、R、Linux、空間的遺伝子発現解析に必要なソフトウェア環境がセットアップされています。 10x Xenium Analayzer発現マトリクスと画像ファイルから、一連の空間的遺伝子発現解析を実行できます。 複数の論文に基づき作成し複数のデータで検証した解析手順をご使用いただけます。
空間的
遺伝子発現解析を
自動化できる解析ソフトウエア
60万円~(税別)
※PC用永続版ライセンスの場合
特徴
・Xeniumを使用した一連の空間的遺伝子発現解析をシンプルなパラメータ設定とクリック操作で実行することが可能です。
・「空間座標の選択」「着目領域の抽出」「抽出した領域に対する解析」を一貫して実行できるため、着目した領域に対する解析を効率的に進めることができます。
・コードを柔軟に書き換えることができるため、配色やレイアウトをカスタマイズすることが可能です。
機能一覧
| 機能 | 詳細 | |
| 1 | QC | QCではUMIカウントや遺伝子数のバイオリンプロットを表示し確認します。解析精度を保つため遺伝子発現の検出数が少ない細胞などを除外します。 |
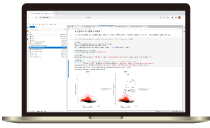
| 2 | 次元削減とクラスタリング | PCAを用いた次元削減と、UMAP、t-SNEを用いたクラスタリングを行い可視化します。また、クラスタ間での発現量の比較解析を行います。 |
| 3 | 細胞種同定 | 各クラスタで高発現な遺伝子を同定し、マーカー遺伝子を探索します。各クラスタで有意に発現している遺伝子から、細胞種同定を行います。 |
| 4 | マーカー遺伝子発現量可視化 | 細胞や遺伝子のクラスタリング結果を空間座標上にマッピングし表示します。どのような細胞が組織切片上のどの位置に分布しているのか、クラスターの境界がどこにあるのかを視覚的に確認することができます。 |
| 5 | 着目領域の抽出 | 着目領域の空間座標を選択して拡大表示することができます。特定の領域に注目することで、局所的な細胞や発現の分布を詳細に確認することができます。 |
tSNE
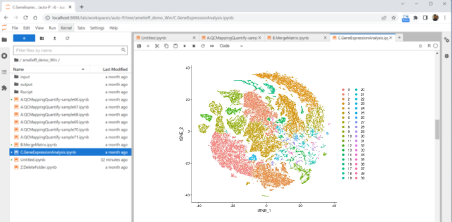
空間プロット
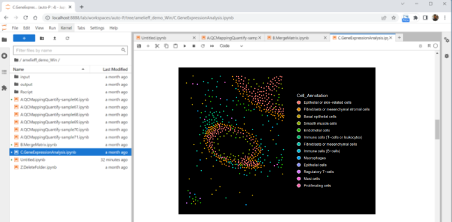
組織図上でのクラスタの可視化
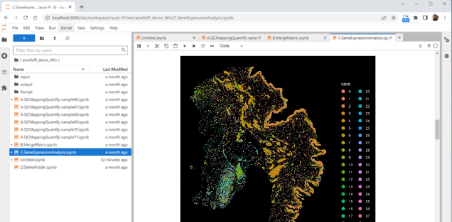
マーカー遺伝子の発現量の可視化




その他
| 必要PCスペック | CPU : 16 core 以上 メモリ : 128 GB 以上 HDD : 2TB 以上 |
| 導入ツール | Seurat, Seaborn, UMAP, t-SNE, jupyter lab, R, Docker |
| 入力データ | 10x Xenium Analayzer 発現マトリクス、画像ファイル |
| 対応生物種 | ヒト、マウス、ラット |

