新年度RNA-seq受託解析キャンペーン

新年度RNA-seq受託解析キャンペーン
ご好評につき期間延長決定!
キャンペーン期間:2022年7月31日 試料またはデータ受領まで
RNA-seq遺伝子発現量群間比較
シーケンスに加えて、論文作成には欠かせない「
二群間解析における発現量比較解析」「
発現変動遺伝子の抽出」「
特定遺伝子に着目した発現量の変化の可視化」「
GO解析」「
パスウェイ解析」込みのスペシャルプラン
RNAseq解析
RNAseq解析とは、次世代シーケンサー(NGS)を用いて転写物の塩基配列を決定する方法です。この配列をリファレンスゲノムへアライメントし、転写産物毎の発現量を測定し、通常はサンプル間の発現量の比較解析を行います。スプライスバリアント毎の発現解析、融合遺伝子検出、変異解析、新たな転写産物の予測を行うことができます。
リファレンスゲノムのない生物の場合でも、配列をアセンブルして転写産物モデルを構築し、アライメントさせて解析することができます。
実験のポイント
解析対象のRNAの種類に適したライブラリー調整キットを用いて、ライブラリー作成を行います。必要なシーケンス量は、生物種や何をプロファイリングしたいかによって異なります。ヒトやマウスの場合、最低でも2000万リード、4Gbp以上の測定が推奨されます。スプライスバリアント(アイソフォーム)の検出や新規転写産物を探索する場合、もしくはFFPEサンプルやnon-conding RNAを扱う場合には、シーケンス量を増やす必要があります。
解析のポイント
NGSによるシーケンスで得られた配列は、FASTQというファイル形式で保存されます。この配列をスプライシングを考慮してゲノム配列にマッピングします。この結果は、BAM形式ファイルで保存され、IGV (Integrative Genomics Viewer)などのゲノムビューワーを用いて閲覧できます。
発現量は、リードカウント、または、補正値を用います。FPKM (Fragments Per Kilobase of exon per Million mapped fragments)やTPM (Transcripts per million)は、発現量をエクソン長と全マッピング数で補正した値です。CPM (Counts per Million mapped reads)は、発現量を全マッピング数で補正した値です。
2群間の比較解析では、発現量の比をlogスケールで表現した値、もしくはt検定による有意差P値がよく用いられます。
研究目的別高次解析のポイント
臨床上の特徴や薬剤が遺伝子発現にどのような影響を及ぼすのかを、発現変動が観測された遺伝子に注目し、特定のパスウェイや生物学的な機能のまとまりで、その意味を捉えることが高次解析の目的です。
このためには以下に示しますように、データの特徴を把握した上で2群間比較による発現変動遺伝子解析を行い、その上で研究目的に応じた生物学的な機能を理解するための解析手法を適切に選択することが重要となります。
データの特徴の把握
・主成分分析
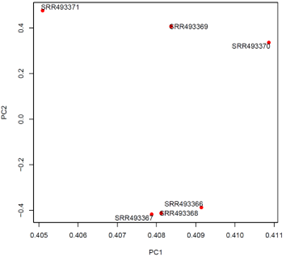
| 全サンプルを対象に、遺伝子発現の傾向を二次元プロットで表現します。これにより、二次元上にプロットされた各サンプル間の距離から、類似性を読み取ることができます。複数サンプルの発現プロファイルの類似度を視覚化し、その後の解析で群間の差を見出すことができるデータであることを確認します。 |  |
・階層的クラスタリングのヒートマップ

|
縦軸に遺伝子、横軸にサンプルを配置し、発現量を色で表現します。 各軸でクラスタリングを実施し、群ごとに同じまたは近いクラスタに分類します。はずれ値を持つサンプルを確認できます。 |
 |
2群間比較による発現変動遺伝子解析
2群間で有意に発現量に差がある遺伝子群を特定します。
・Volcano plot
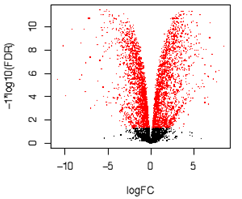
|
2群間の比較解析において、有意差P値(logスケール)と発現量の比(logスケール)を2次元プロットで表現します。各プロットの点は遺伝子に該当し、有意に変動している(p値や発現量比)遺伝子数の概観の確認や、変動の傾向の把握に使用します。 各点に該当する遺伝子名を重ねて表記するなどし、既知の遺伝子で想定される発現変動が起きているかを確認するなど、データの妥当性の検討に用います。 |
 |
発現変動遺伝子と生物学的意義
発現変動した遺伝子群の生物学的機能の共通点や、影響を受けているパスウェイ等の探索を行います
・GO(Gene Ontology)解析
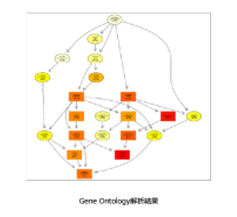
|
Gene Ontologyとは、遺伝子の属性を説明する語彙を体系化・構造化した記述方法です。GOは生物学的プロセス、細胞の構成要素、分子機能の3カテゴリーに分けることができ、それぞれのカテゴリーにはさらなる下位概念が定義されています。有意に変動した遺伝子群がどのような生物学的概念にエンリッチしているかを解析することで、比較対象が影響を与えている生物学的機能を捉えます。 最上位GOから、下位GOまでの繋がりを図に示します。それぞれのGOのエンリッチメント解析結果は色の濃淡で有意差(P値)の大きさを表します。 |
 |
アメリエフが選ばれる理由
- ・実験と解析をワンパッケージでご提供します
- ・研究目的にあった解析手法を選択し解析プランを組み立てます
- ・解析内容と解析結果を丁寧にご説明し仮説検証に貢献します
解析メニュー
- RNAシーケンス
- ライブラリ調製、NovaSeq6000による(PolyA 4Gb/13.3Mぺアードリード)によるシーケンシング
-
- バイオインフォマティクス
- 解析レポート、中間報告、最終報告(手法解説、結果解釈のディスカッション)
- 発現定量解析:マッピング、 発現定量、クラスタリング、ヒートマップ、20サンプル以上の場合は主成分分析
- 二群間解析における発現量比較解析:二群間解析(Volcano plot、4組組み合わせまで)、クラスタリング、ヒートマップ、 主成分分析
- 発現変動遺伝子の抽出
- 特定遺伝子に着目した発現量の変化の可視化(Box plotなど)
- GO解析(Gene Ontology)、パスウェイ解析、20サンプル以上の場合はWGCNA(重み付け遺伝子共発現ネットワーク解析)
- ※non stranded RNA-seqとなります。strand specific RNA-seqをご希望の場合はお問合せください
- ※12Gbの場合(NovaSeq6000によるPolyA 12Gb/40Mぺアードリード)、6.9万円/サンプル(税込75,900円)となります。
サンプルに関して
サンプル要件
- RNA-seq遺伝子発現量群間比較
- サンプルタイプ:total RNA
※サンプルQCでRIN値が非常に低い場合は、再提出をお願いすることがございます。
※ ヒトおよびマウス以外の生物種をご希望の場合は、お問合せください。総量(µg) 液量(µL) 濃度(µg) 純度 1以上 20以上 50以上 OD260/280 1.8 – 2.2
OD260/230 ≧ 1.8
- サンプルタイプ:total RNA
送付方法
サンプルは、ドライアイス同梱の上、ノボジーン東京配送センターにお送りください。
日本橋ライフサイエンスビルB101 東京配送センター
注意事項
- ※シーケンスデータはノボジーン株式会社が提供いたします
- ※東京配送センター利用料金、データ納品のHDD/Cloudの費用は別途頂戴いたします。
- ※シングルセル遺伝子発現解析は、機器を持ち込んでの出張サービスも承っております。
その際には出張費をご請求いたします。
また、ノボジーンラボにサンプルを持ち込んで頂いてのご利用も可能です。詳細はお問合せください。