WESがん変異解析|WET&DRY【新製品発売記念キャンペーン】

シーケンスに加えて、
論文作成には欠かせない
データ解析・作図まで込みの
スペシャルプライス
14.9万円/症例
(税込163,900円)
サービス内容
【Whole Exome シーケンス】| ○ ライブラリ調製 |
| ○ 腫瘍組織 12Gb/サンプル(NovaSeq6000 PE150) |
| ○ 正常組織 6Gb/サンプル(NovaSeq6000 PE150) |
| ○ 解析レポート、最終報告(手法解説、結果解釈のディスカッション) |
| ○ 体細胞変異検出:クオリティコントロール、マッピング、体細胞変異(SNV/InDel)検出、遺伝子情報アノテーション |
| ○ データベースアノテーション及び計算予測:dbSNP、gnomAD、ClinVar、PROVEAN、SIFT、phastCons |
| ○ 高次解析(3症例, 6サンプル以上の場合):Oncoplot作成、Tumor Mutation Burden 算出、Driver gene解析 |
| ※サンプル数によって対応可能な組み合わせ数が異なりますのでご相談ください |
サンプル要件 / 注意事項
サンプルタイプ:genomic DNA
| 推奨DNA量(ng) | 液量(µL) | 濃度(ng/µL) | 純度 |
|---|---|---|---|
| 300以上 | 20以上 | 20以上 | OD260/280 ≥ 1.8 OD260/230 ≥ 1.8 no degradation, no contamination |
- ※ 過度のRNAの混入がないこと。タンパク質・フェノール等の不純物の混入がないこと。
- ※ 再懸濁溶液は、純水 ( DNase/RNase-free )、EBまたはlow TE ( < 0.1 mM EDTA )
- ※ シーケンシングは海外(シンガポール)にて実施します。オプションとして国内でのシーケンシングも対応可能です。
- ※ 海外輸送費・ 納品にかかる費用 (HDD) を別途頂戴します。
- ※ 対象生物種はヒトのみとなります。
ご利用の流れ
- 研究目的、サンプル情報をヒアリングし、お見積りを作成します。
- ご発注後、サンプルを準備いただき、シーケンスを行います。
- 報告会にて、高次解析結果の報告および解釈について、ご報告します。
WESがん変異解析とは
がんの診断や治療薬の効果・副作用の予測、治療抵抗性や予後の予測において、ゲノム変異がバイオマーカーとして活用されつつあります。特に体細胞変異は治療薬の効果を予測するマーカーとなり得ます。近年急速にゲノム配列のシーケンシングコストが低下しており、マーカーを探索するために全ての遺伝子領域における変異を網羅的に解析するホールエクソームシーケンス(WES)を活用することが一つの有効な手段となっています。
がんと正常サンプルのシーケンスデータを比較することで体細胞変異を検出し、既知のアノテーションを付与し、変異が蓄積している遺伝子と臨床情報の関連性を可視化するなどの解析を通じて、バイオマーカーの候補を検討することができます。
実験のポイント
一般的には、がん変異解析には同一患者のがん組織と正常組織のゲノムDNAが必要となるため、それぞれの組織のサンプルを準備します。がん組織のゲノムDNAは、手術検体試料や病理診断の際の生検試料の一部をサンプルとして利用することが多いため、ホルマリン固定パラフィン包埋(FFPE)組織サンプルからの抽出が主となります。ただし、FFPEの検体は試料作成までの時間によってDNAの品質の低下をもたらす可能性があるため、組織抽出から固定までの時間を短くする必要があります。また、ホルマリン固定液の組成、濃度、pHなどの条件も重要となります。さらに、ホルマリンに長期固定すると、DNAの塩基置換が生じて、それが変異の擬陽性として検出されます。
一方、新鮮凍結(FF)サンプルは、DNA抽出に非常に適していますが、サンプル調製と保存に費用がかかります。
次世代シーケンサ(NGS)でシーケンスする際に、採取したがん組織のサンプルに多くの正常細胞が混入していると、がん細胞由来のゲノムDNAの割合が相対的に減少するため、検出精度の低下を招く可能性があり、米国がんゲノム解析プロジェクト等ではがん細胞含有率は30%以上であることが推奨されてます。
一方、正常組織のゲノムDNA の採取は、おもに末梢血から密度勾配遠心法を用いて分離された単球やリンパ球を含む単核球(PBMC :Peripheral Blood Mononuclear Cells)より採取されます。
がんのWESでは、がん組織のゲノムを高解像度で決定する必要があるため、がん組織のサンプルを約 x100(12Gb)、正常組織のサンプルを 約 x50(6Gb)程度のカバレッジでシーケンスするなど、がん組織のサンプルを厚めにシーケンスすることが推奨されています。
変異解析のポイント
NGSによるシーケンスで得られた配列は、FASTQというファイル形式で保存されます。この配列を参照ゲノム配列にマッピングします。この結果は、BAM形式ファイルで保存され、IGV (Integrative Genomics Viewer)などのゲノムブラウザを用いて可視化できます。
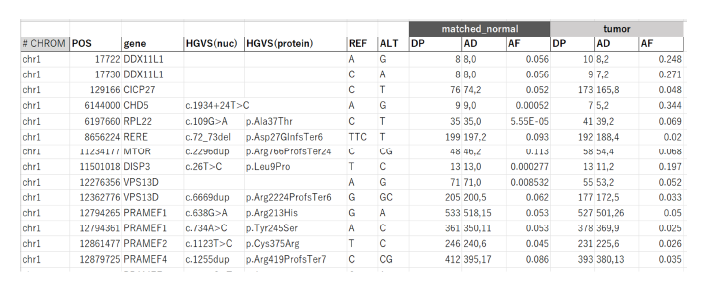
腫瘍組織のシーケンスデータには、生殖細胞系列の変異も含まれているため、正常組織のシーケンスデータと比較し、生殖細胞系列の変異を除いた体細胞変異を検出します。その際は、正常組織の混入により腫瘍組織の純度は100%でないことや、腫瘍組織に複数のサブクローンが存在すること、コピー数変異が存在することを考慮し、それぞれの変異について、アレルの割合(variant allele frequency, VAF)の変動を許容して体細胞変異を検出します。
検出された変異は、どの遺伝子上の変異か、どの程度翻訳後のアミノ酸に影響を与えるかなどの生物学的なアノテーション(意味づけ)、疾患との関連が報告されているかなど公共データベースと照らし合わせる方法でナレッジベースの分析を行い、着目すべき変異を絞り込みます。
【変異抽出結果の例】
・IGVによる変異の可視化
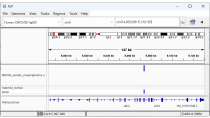
| 解析によって得られたマッピング情報(BAM形式)と体細胞変異情報(VCF形式)は、フリーの可視化ツールであるIGVに読み込ませることで、シーケンスの様子と候補となる変異を視覚的に把握することができます。 特に、腫瘍組織割合やサブクローンの影響によって生じた低VAFの様子を直感的に理解するために役立ちます。 |  |
高次解析のポイント
・Oncoplot(オンコプロット)

| 遺伝子ごとに観測された変異の種類、サンプル全体に占める割合を要約・可視化することで、患者群に共通する遺伝子や変異の特徴を視覚的に把握することができます。さらに、遺伝子変異を臨床情報のカテゴリごとに分けてプロットすることで、臨床的な特徴と関連している特定の遺伝子変異の有無を探索することができます。 |  |
・TMB解析(Tumor Mutation Burden 解析)
遺伝子変異量(TMB)とは、がん細胞に蓄積した体細胞変異の総量を示す値で、百万塩基あたりの変異塩基数で表されます。TMBは、がんの種類や発生メカニズムにより特徴的になることが知られているほか、免疫チェックポイント阻害剤の治療効果予測にも用いられています。
・Driver gene解析
発がんやがんの悪性化の直接的な原因となるような遺伝子をドライバー遺伝子と呼びます。既知のドライバー遺伝子に変異が存在するかを解析することで、がんの原因遺伝子を推測することができます。既知の遺伝子として、TCGA(The Cancer Genome Atlas)など公共データベースで取りまとめられているリストを用います。

